Modèle ruban
Dans cet article, nous allons approfondir le sujet de Modèle ruban et explorer ses nombreuses facettes. Modèle ruban est un sujet qui a suscité un grand intérêt dans divers domaines, de la science à la culture populaire. Tout au long de l’histoire, Modèle ruban a fait l’objet de débats, de recherches et de réflexions, et reste d’actualité aujourd’hui. Nous approfondirons les différentes perspectives qui existent sur Modèle ruban, en abordant son importance, ses implications et son impact sur la société. A travers cet article, nous souhaitons proposer une vision complète et actualisée de Modèle ruban, dans le but d’enrichir les connaissances et de favoriser la réflexion sur ce sujet passionnant.
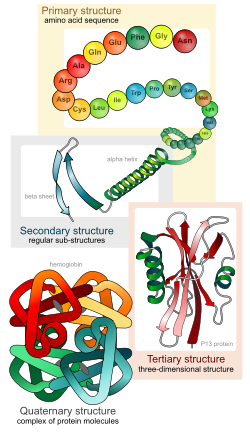
Le modèle ruban est une modélisation moléculaire de la structures des protéines[1],[2],[3],[4]. Un ruban permet de visualisr des éléments de la structure secondaire comme une hélice alpha ou un feuillet bêta et permet de représenter la structure tertiaire. Les divers peptides peuvent être distingués par des couleurs. Les modèles ruban sont utilisés en modélisation moléculaire[5],[6] par exemple en ingénierie des protéines (de). Le modèle a été établi par Jane Richardson en 1980/1981[1],[2],[3].
Conventions


| Structure secondaire[1],[2] | |
|---|---|
| Hélice alpha | Ruban cylindrique en spirale, le plan du ruban se trouvant approximativement au niveau de la liaison peptidique. |
| Feuillet bêta | Flèche d'une épaisseur de 25 % de la largeur, dirigés de l'extrémité N-terminale vers l'extrémité C-terminale. Les feuillets bêta sont constitués de brins β et de boucles β. |
| Brins et autres | |
| Brins non répétitifs | Brins ronds le long des atomes α C des acides aminés, qui s'amincissent du premier plan vers l'arrière-plan. |
| Connexions entre boucles et hélices | Brins ronds le long des atomes α C des acides aminés, qui s'aplatissent lors du passage à l'hélice alpha |
| Autres caractéristiques | |
| Extrémités N-terminale et C-terminale | Petites flèches éventuellement avec lettres. Pour les brins β, la direction de la flèche suffit ; un dégradé de couleurs le long de la séquence d'acides aminés est souvent utilisé à cette fin. |
| Ponts disulfure | Avec un double S entrelacé ou avec une ligne en zigzag |
| Groupes prothétiques ou inhibiteurs | Modèle en bâton ou en boule |
| Métaux | Boules |
| Ombres et couleurs | Le contraste diminue vers l'arrière-plan |
Logiciels
Divers programmes ont été développés pour la représentation des protéines dans le modèle ruban, par exemple Molscript de Arthur M. Lesk (en), Karl Hardman et John Priestle[7], Jmol[8], DeepView[9], MolMol[10], KiNG[11], UCSF Chimera (en)[12] ou PyMOL[13].
Liens externes
Notes et références
- (de) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en allemand intitulé « Bändermodell (Proteine) » (voir la liste des auteurs).
- Jane S. Richardson, « The anatomy and taxonomy of protein structure », Advances in protein chemistry, vol. 34, , p. 167–339 (PMID 7020376).
- Jane S. Richardson, « Schematic drawings of protein structures », Methods in enzymology, vol. 115, , p. 359–38 (PMID 3853075).
- Jane S. Richardson, « Early ribbon drawings of proteins », Nature Structural & Molecular Biology, vol. 7, no 8, , p. 624–625 (PMID 10932243, DOI 10.1038/77912, lire en ligne).
- ↑ M. Morange, « What history tells us XXV. Construction of the ribbon model of proteins (1981). The contribution of Jane Richardson. », Journal of Biosciences (de), vol. 36, no 4, , p. 571–574 (PMID 21857104, lire en ligne).
- ↑ Peter D. Sun, Christine E. Foster et Jeffrey C. Boyington, « Overview of Protein Structural and Functional Folds », Current Protocols in Protein Science, vol. 35, no 1, (PMID 18429251, DOI 10.1002/0471140864.ps1701s35).
- ↑ Mike Carson et Charles E Bugg, « Algorithm for ribbon models of proteins », Journal of Molecular Graphics, vol. 4, no 2, , p. 121–122 (DOI 10.1016/0263-7855(86)80010-8).
- ↑ P. J. Kraulis, « MOLSCRIPT: a program to produce both detailed and schematic plots of protein structures », Journal of Applied Crystallography, vol. 24, no 5, , p. 946–950 (DOI 10.1107/S0021889891004399).
- ↑ Angel Herráez, « Biomolecules in the computer: Jmol to the rescue », Biochemistry and Molecular Biology Education, vol. 34, no 4, , p. 255–261 (PMID 21638687, DOI 10.1002/bmb.2006.494034042644).
- ↑ Maria U Johansson, Vincent Zoete, Olivier Michielin et Nicolas Guex, « Defining and searching for structural motifs using DeepView/Swiss-PdbViewer », BMC Bioinformatics, vol. 13, no 1, , p. 173 (PMID 22823337, PMCID 3436773, DOI 10.1186/1471-2105-13-173).
- ↑ Reto Koradi, Martin Billeter et Kurt Wüthrich, « MOLMOL: A program for display and analysis of macromolecular structures », Journal of Molecular Graphics, vol. 14, no 1, , p. 51–55 (PMID 8744573, DOI 10.1016/0263-7855(96)00009-4)
- ↑ Vincent B. Chen, Ian W. Davis et David C. Richardson, « KING (Kinemage, Next Generation): A versatile interactive molecular and scientific visualization program », Protein Science, vol. 18, no 11, , p. 2403–2409 (PMID 19768809, PMCID 2788294, DOI 10.1002/pro.250).
- ↑ Thomas D. Goddard, Conrad C. Huang et Thomas E. Ferrin, « Software Extensions to UCSF Chimera for Interactive Visualization of Large Molecular Assemblies », Structure, vol. 13, no 3, , p. 473–482 (PMID 15766548, DOI 10.1016/j.str.2005.01.006).
- ↑ Axel T Brunger et James A Wells, « Warren L. DeLano 21 June 1972–3 November 2009 », Nature Structural & Molecular Biology, vol. 16, no 12, , p. 1202–1203 (PMID 19956203, DOI 10.1038/nsmb1209-1202).